バイオインフォマティクス分野

2008年 東京大学大学院 農学生命科学研究科 応用生命化学専攻博士課程修了
2008年 京都大学 化学研究所附属バイオインフォマティクスセンター 特定研究員
2010年 科学技術振興機構 さきがけ 専任研究員
2015年 医薬基盤・健康・栄養研究所 プロジェクト研究員
2021年 医薬基盤・健康・栄養研究所 プロジェクトリーダー(現職)
2022年 徳島大学 先端酵素学研究所 特任教授(現職)
2025年 医薬基盤・健康・栄養研究所 AI健康・医薬研究センター センター長(現職)
研究概要
バイオインフォマティクス分野では、AI(人工知能)/ ML(機械学習)の手法を用いて診療情報やオミックスデータ(生体分子を網羅的に測定したデータ)の解析を行うことによって創薬支援につなげることを目指した研究を行っています。また、創薬支援を目的としたデータベースの構築及びデータベース構築そのものを支援するプラットフォームの開発を進めています。
データ駆動的な患者層別化と創薬標的探索
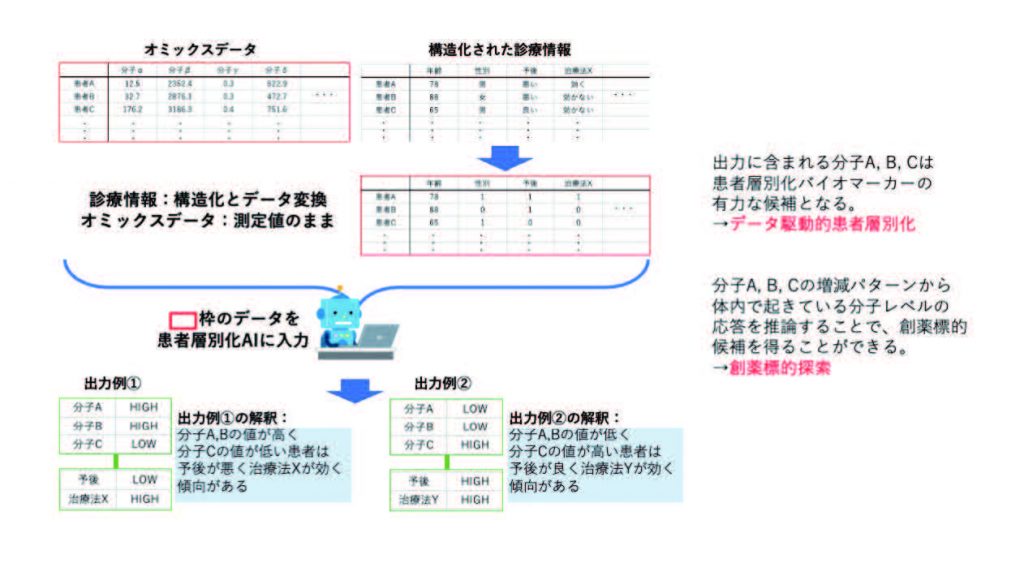
診療情報とオミックスデータを紐づけて解析するための解析手法を開発し、病気のフェノタイプとの関連が示唆される生体分子を検出することによって、患者層別化を行うアプローチを進めています。診療情報は、はい/いいえなどのカテゴリ変数で記載される項目や強度などを示す数値(離散変数)、測定結果などを示す数値(連続変数)など異なるタイプの項目が混在する傾向があります。一方、オミックスデータは測定結果であれば連続変数の行列として取得されることから、診療情報とオミックスデータを各項目のデータタイプに依存することなく紐付けて解析するためにはどのような解析を行えば良いかを考える必要があります。当研究室で開発された解析手法を用いて診療項目と生体分子の連関を出力し、その結果を介して患者層別化を実施することにより、共通点のある患者グループにおいてどのような生体分子が創薬標的になりうるかをデータ駆動的に提示することができます。
各種データベースの構築支援
創薬支援を目的として収集された大規模なオミックスデータセットを格納したデータベースや、複数の研究機関がそれぞれ独自に収集したデータを一拠点で収集・管理するためのデータベース構築を進めています。その他、メタデータを付随させたオミックスデータのデータベースを簡便に構築できるようにするプラットフォームの開発を行っています。
オミックスデータを用いた化合物の毒性発現メカニズム推定
化合物を実験動物に投与した際に得られる遺伝子発現プロファイルを元にして、毒性発現にどのような生体イベントが関与しているのかを既存データベースやバイオインフォマティクスツールを用いて推定するパイプライン構築を行っています。
最近の主要論文
- Enomoto, T., Shirai, Y., Takeda, Y., Edahiro, R., Shichino, S., Nakayama, M., …, Natsume-Kitatani, Y., …, & Kumanogoh, A. (2024). SFTPB in serum extracellular vesicles as a biomarker of progressive pulmonary fibrosis. JCI insight, 9(11), e177937.
- Tomoto, M., Mineharu, Y., Sato, N., Tamada, Y., Nogami-Itoh, M., Kuroda, M., …, Natsume-Kitatani, Y. & Okuno, Y. (2024). Idiopathic pulmonary fibrosis-specific Bayesian network integrating extracellular vesicle proteome and clinical information. Scientific Reports, 14(1), 1315.
- Haga, A., Iwasawa, T., Misumi, T., Okudela, K., Oda, T., Kitamura, H., …, Natsume-Kitatani, Y., …, & Ogura, T. (2024). Correlation of CT-based radiomics analysis with pathological cellular infiltration in fibrosing interstitial lung diseases. Japanese Journal of Radiology, 42(10), 1157-1167.
- Allendes Osorio, R. S., Kosugi, Y., Nyström-Persson, J. T., Mizuguchi, K., & Natsume-Kitatani, Y. (2024). A modern multi-omics data exploration experience with Panomicon. Bioinformatics Advances, 4(1), vbae147.








